Si Anfinsen avait su...
Aujourd’hui, au menu : génétique. Avec un chapitre de l’histoire des plolymorphismes mononucléotidiques ou single nucleotide polymorphisms (SNPs), chapitre qui risque de faire du bruit. Déjà, l’article est paru dans Science (1), ce qui est un bon début question gestion d’image.
Les SNP sont des variations de la séquence d’un gène touchant un seul nucléotide, plus ou moins fréquentes dans la population générale, et n’ayant « en principe » pas de conséquence fonctionnelle. Il y en aurait environ 1/10000 paires de base dans le génome.
On sait bien aujourd’hui qu’un grand nombre de maladie sont multifactorielles, et admettent des facteurs environnementaux et génétiques. Le facteur génétique est alors le plus souvent polygénique. Ainsi, la modification d’un seul gène n’est pas suffisante pour le développement de la maladie. Le mode de transmission de maladies, et plus largement de phénotypes polygéniques, n’est donc pas mendélien. Pour les étudier, on peut rechercher une association (et pas une liaison) entre le phénotype et certains polymorphismes, par exemple de type SNP. (Ca va jusqu’ici ?)
Ces dernières années, plusieurs associations ont ainsi été mises en évidence. L’une d’entre elle concerne des SNP du gène MDR1 (Multi-drug resistance, le nom importe peu) codant pour une protéine PgP (idem) qui est un transporteur multidrogue : elle fait sortir plein de molécules de la cellule (genre vous quand vous êtes de corvée poubellistique). Des modulations de son activité pourraient expliquer des phénomènes de résistance à divers traitements. Des études d’association ont obtenu des résultats peu reproductibles (c’est leur problème principal) et parfois contradictoires (gain ou perte de fonction). Et de toutes façons, une association, c’est bien joli, mais ça ne signifie pas causalité.
L’étape suivante est donc de démontrer que le SNP a un retentissement fonctionnel (2). C’est l’objet de l’article sus-cité.
Le polymorphisme en question, que j’appellerai A par commodité, est dit synonyme : le codon mutant code pour le même acide aminé. Mais il est en fait transmis en bloc avec deux autres SNPs (= haplotype), l’un B synonyme aussi , l’autre C non-synonyme. Les auteurs ont construits plein de mutants avec toutes les combinaisons possibles de ces 3 SNP : isolés, par paire, puis les 3, et les ont transfectées dans plusieurs types de cellules (humaines et simiesques). Ils ont ensuite étudié les effets de divers inhibiteurs de la PgP, et ont montré que la sensibilité de la protéine à leur action était à la présence de A, et non de C, ce qui aurait été « plus commode » puisque C est non synonyme. Par ailleurs, A seul n’était pas suffisant : il faut qu’ils soit associé à B et/ou à C.
Ensuite, ils ont montré:
1) que l’action des inhibiteurs de la PgP étaient moindre sur les cellules mutantes.
2) que ceci n’était pas lié à une diminution de la formation de l’mARN de PgP, ni à la formation d’un mARN plus court que la normale ou de séquence différente de celle "attendue" de par la séquence ADN
3) mais à une conformation tridimentionnelle différente de la protéine. L’hypothèse est la suivante: les codons synonymes ne sont pas équivalents : leur fréquence d'utilisation varie au sein du génome. Or la cinétique de l’activité des ribosomes qui traduisent l’mARN en protéine, n'est pas uniforme. Elle est notamment modulée par la quantité de tARN environnante. Et celle-ci dans de nombreux organismes, est proportionnelle à la fréquence d'usage du codon. Or de la cinétique des ribosomees vont dépendre les interactions entre différentes régions de la protéine au cours de sa synthèse, et donc sa conformation finale. Et ainsi sa fonction. La démonstration de cette hypothèse in vivo est bien compliquée. Mais ce papier in vitro apporte en tous cas une contribution plus que non négligeable (c'est pas très français, ça).
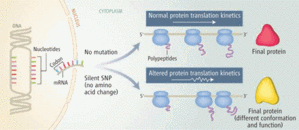
Translation kinetics and protein folding.(3)
Démonstration du caractère éronné d'un vieux principe. Anfinsen écrivit ainsi "the information necessary to specify the native three-dimensional structure of a protein—which largely defines its function—is contained solely in its amino acid sequence" (Science, 1973). Mais bon, c'était il y a 34 ans...
C’est aussi un papier vraiment intéressant, pour les raisons (2). Il y a certes encore du boulot, avant d'expliquer en quoi la résitance au traitement peut être prédite per les SNP du gène MDR1. En effet dans leur papier, si j'ai bien compris, les cellules mutantes avaient la même activité de base vis-à-vis des substrats de PgP que les Wild Type. Seule leur modulation par les inhibiteurs de celle-ci était diminuée. Mais à chaque jour suffit sa peine, n'est-il pas?
(1)A "Silent" Polymorphism in the MDR1 Gene changes Substrate Specificity. C. Kimchi-Sarfaty et al, Science 315, 525 (2007);
(3) SNPs, Silent But Not Invisible. Anton A. Komar. Science 315, 466 (2007)
